説明文の1
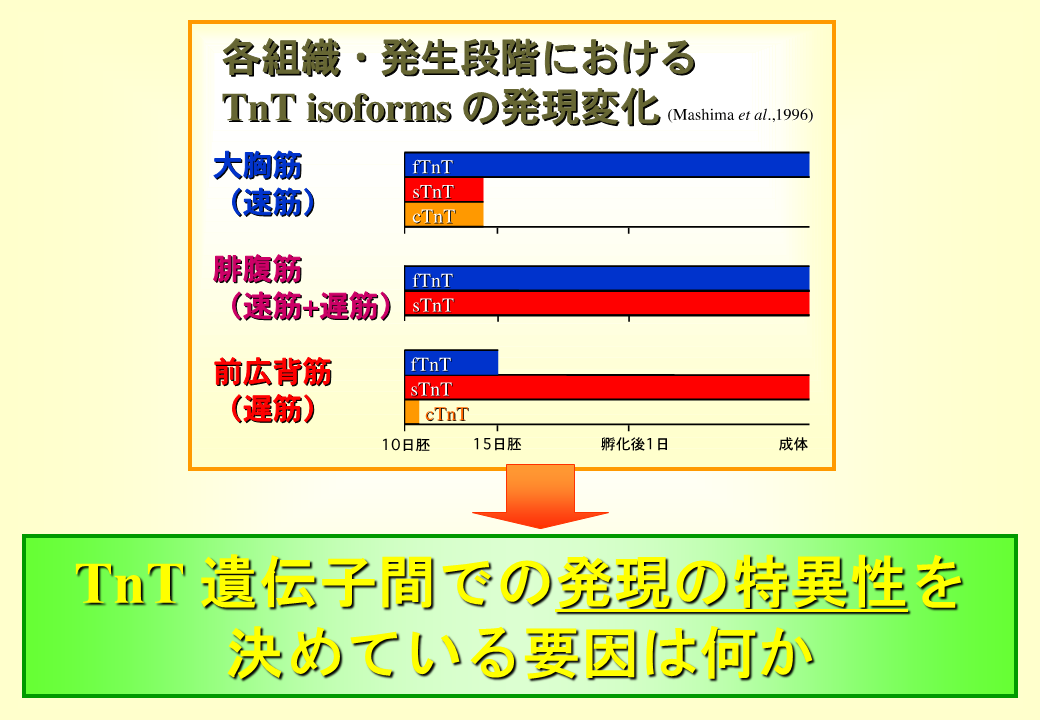
速筋型、遅筋型、心筋型トロポニンT遺伝子の発現は孵化前後までは厳密に調節されていないが、発生が進むに連れて発生段階特異的、筋肉特異的に調節される。孵化前には筋肉の働きはそれほど重要でないかもしれないが、孵化後には各々の役割をしっかり果たさなければならない。速筋は外的な要因が関与しなくても速筋になるので、速筋型トロポニンT遺伝子の発現もそれに同調するのだろう。では遅筋型遺伝子の発現はどうか?
説明文の2
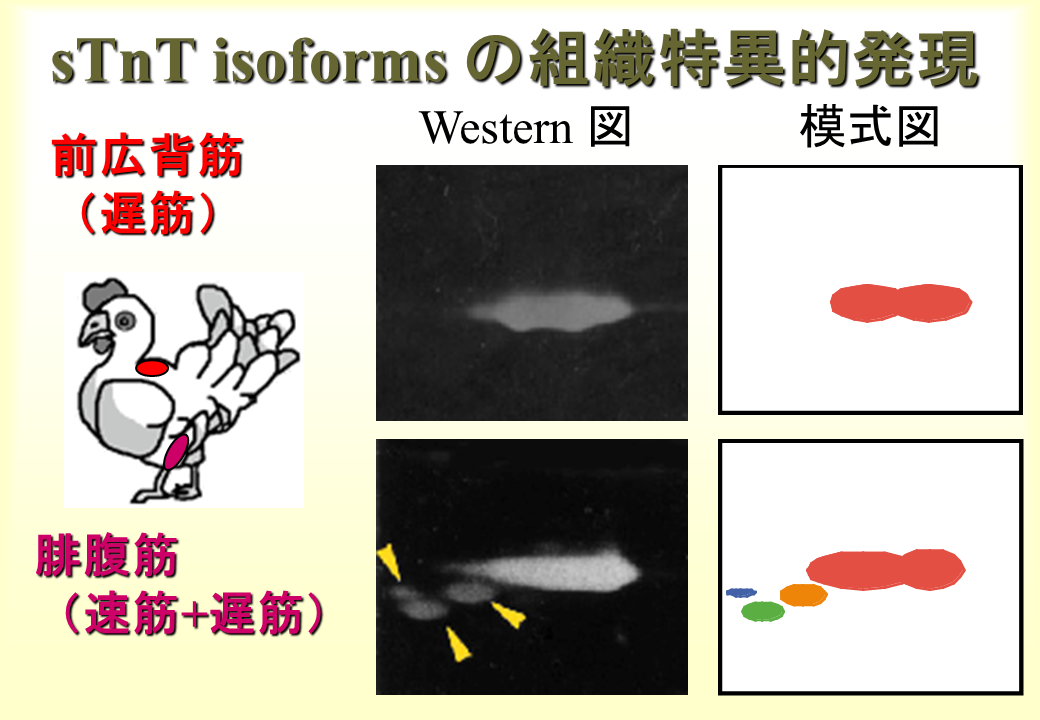
2次元電気移動パターン上で遅筋型トロポニンTは、ほぼ100%遅筋である前広背筋で2つのスポットとして現れる。混合筋である腓腹筋では、よく観察すると、5つスポットとして現れる。後で示すように遅筋型遺伝子には、選択的スプライシングを受けるエクソンが1個しかない。それゆえ2つのアイソフォームが産み出される可能性がある。2つ以上スポットがみられるのは、翻訳後の修飾(リン酸化)のためである。
説明文の3
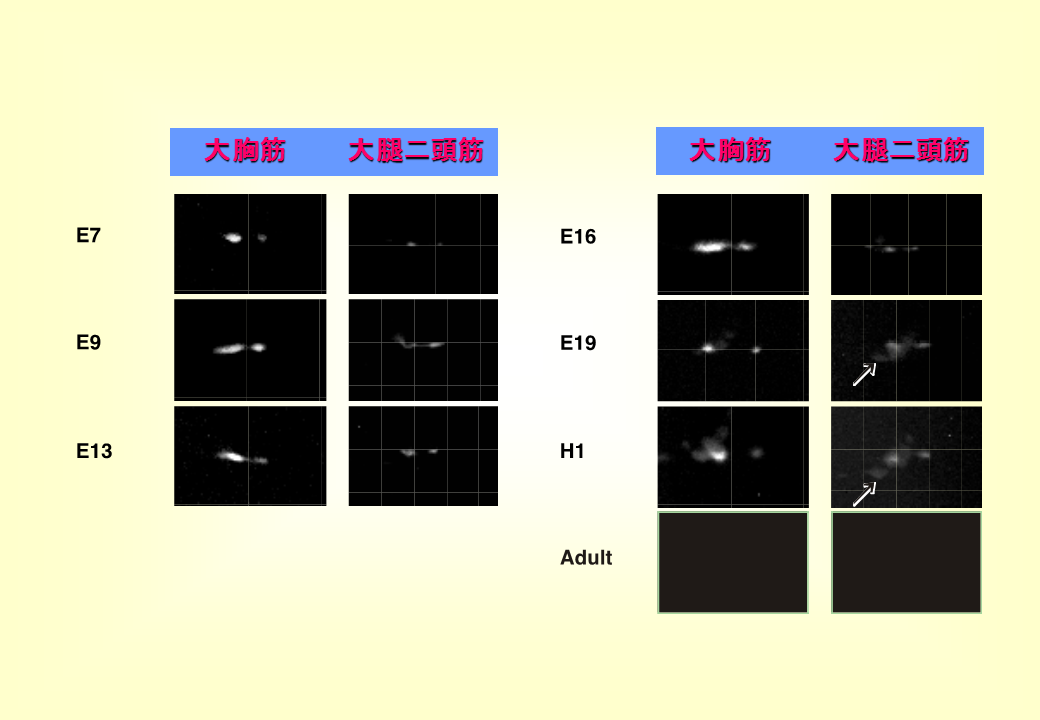
速筋である大胸筋でも、大腿二頭筋でも孵化前後までは遅筋型トロポニンTがみられる。

説明文の4
混合筋や遅筋では、孵化後も遅筋型トロポニンTは発現し続ける。発生段階によって、また筋肉によってみられるスポットの数は異なる。
説明文の5
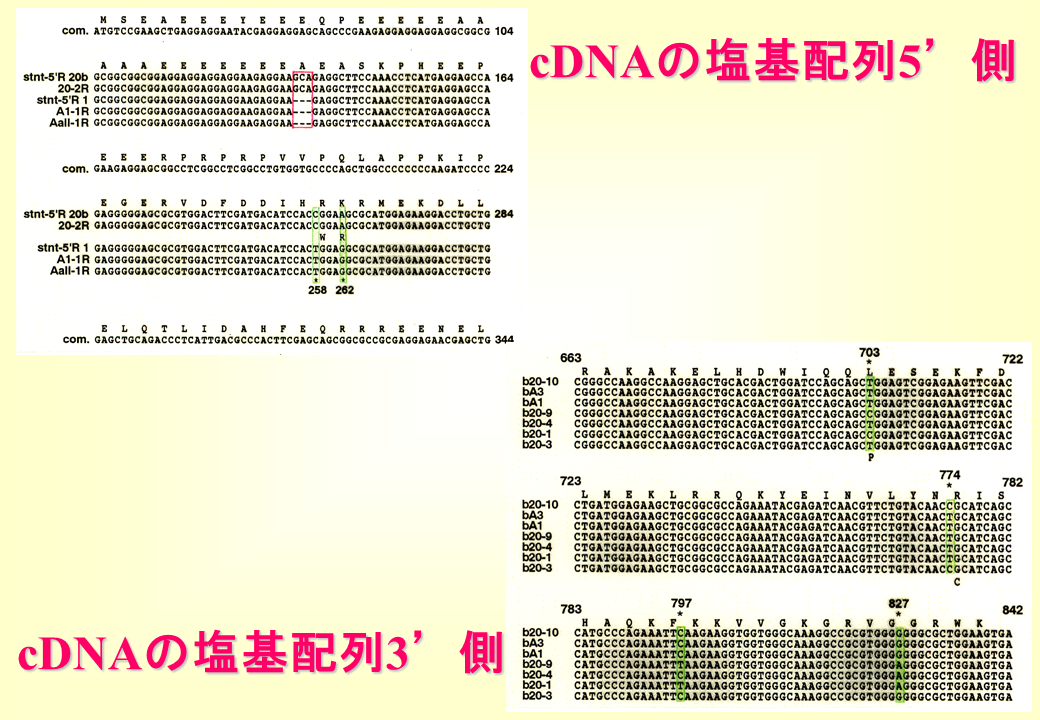
遅筋型トロポニンTのmRNAの(mRNAは不安定なのでcDNAにして)塩基配列を決めたところ、個体によって数カ所で違いがみられた。
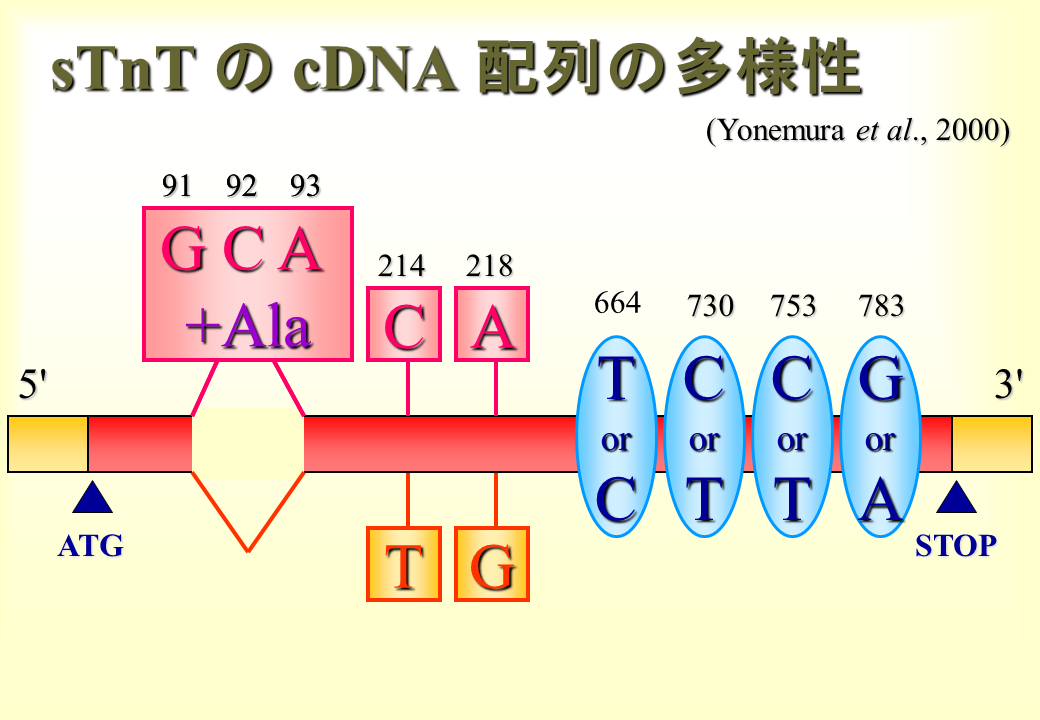
説明文の6
アミノ酸のアラニンを指定するGCAを含む場合と含まない場合があった。この他に6カ所で個体によって塩基配列が違っていた。GCAを含む場合は、214番目はC、216番目はAであった。GCAを含まない場合は、214番目はT、216番目はGであった。
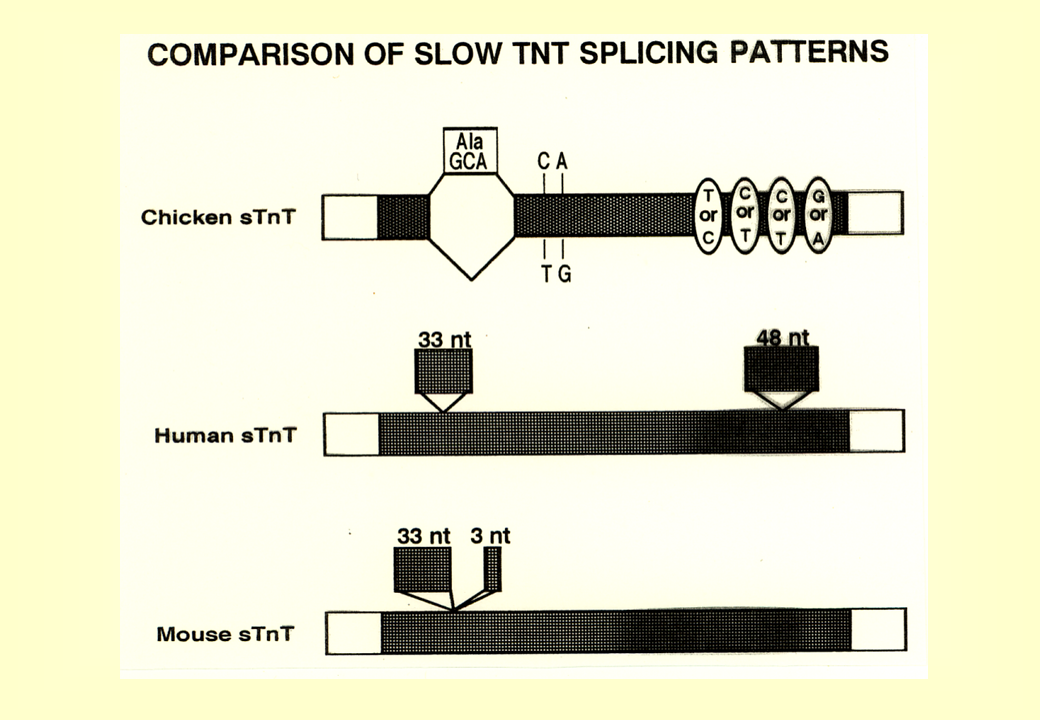
説明文の7
トロポニンTのmRNAでは、ニワトリの場合アラニンを指定するGCAがある場合とない場合があったが、ヒトでもマウスでも特定の塩基がある場合とない場合があった。そのような配列の長さや位置は生物種によって違っていた。
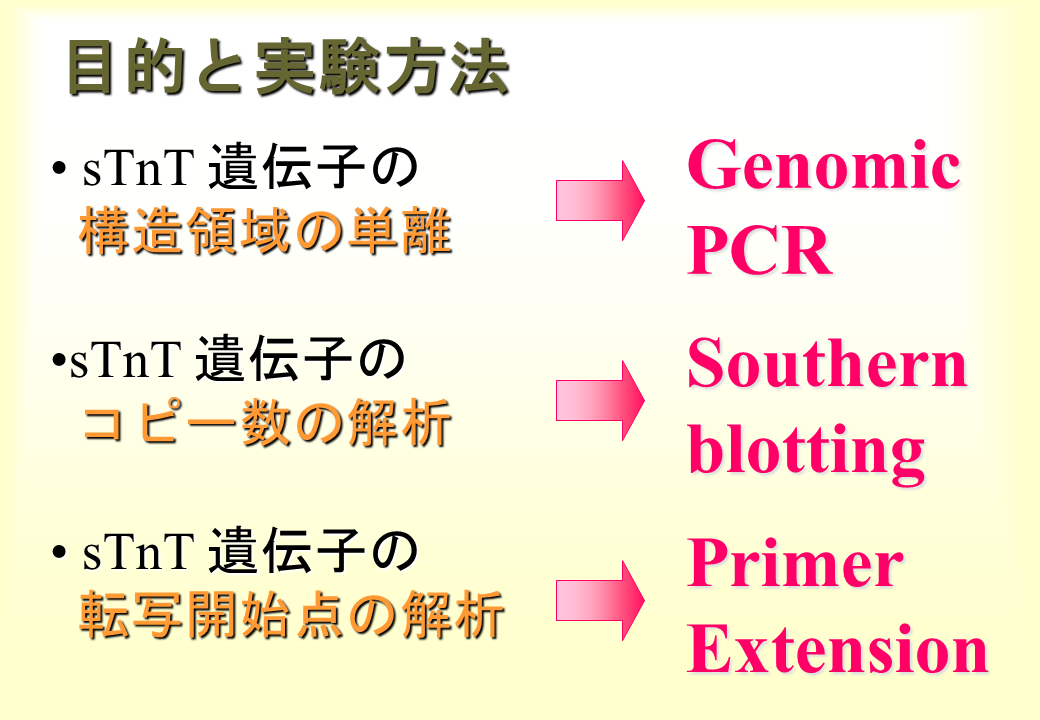
説明文の8
遅筋型トロポニンT遺伝子の構造を決めるためにGenomic PCRを、遅筋型遺伝子の数を決めるためにSouthern blottingを、遺伝子の端(mRNAの始まり)を調べるためにPrimer Extensionをおこなった。

説明文の9
ニワトリの肝臓よりDNAを抽出し、mRNAの塩基配列を基に構築したプライマーSP1とAP1を用いてPCRを行って、遅筋型TnTの塩基配列を増幅した。
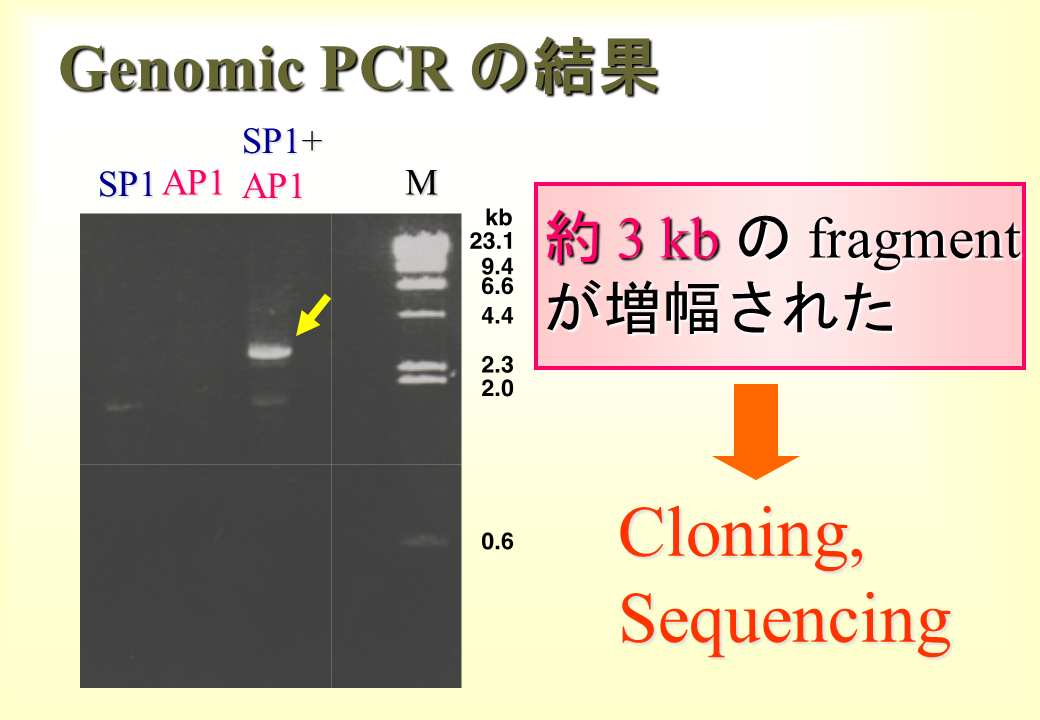
説明文の10
なんと約3 kbのバンドがみられた。速筋型遺伝子の約1/10、心筋型遺伝子の約1/3の長さしかなかった。

説明文の11
遅筋型トロポニンT遺伝子の塩基配列を決定したところ、全長は3155 bpであり、14個のエクソンを含んでいた。
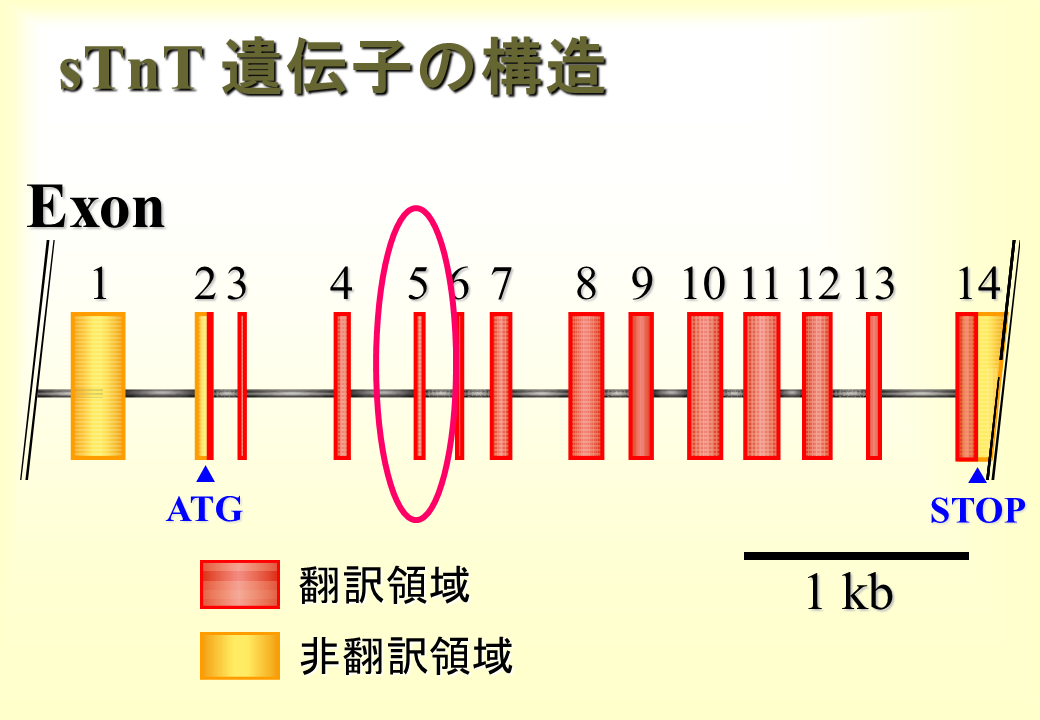
説明文の12
エクソン5が選択的スプライシングを受けることがわかった。
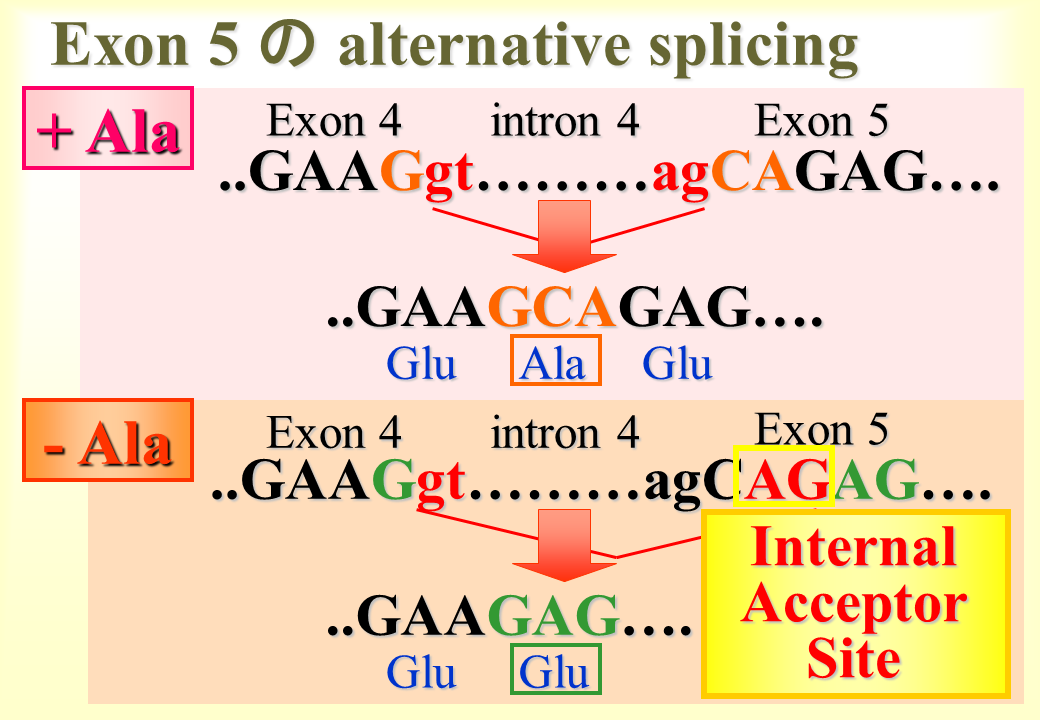
説明文の13
エクソン4とエクソン5がそのままつながると、エクソン4の最後のGとエクソン5の最初のCAによって、アラニンを指定するGCAが含まれることがわかる。しかし、エクソン5の直前のsplice siteであるagを用いずに、エクソン5の2番目のAと3番目のGをsplice siteとして用いると、GCAを含まなくなる。つまり、エクソン5は選択的3‘スプライス部位をもつエクソンなのである。他の6カ所の塩基の相違は選択的スプライシングによるという証拠はなく、個体差による。
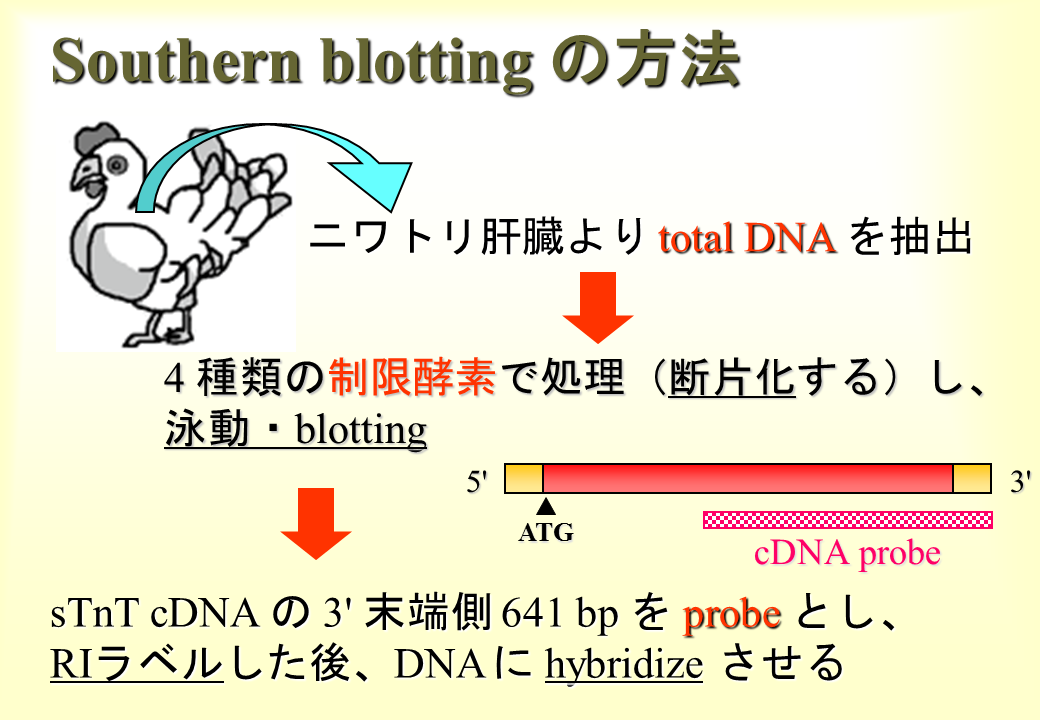
説明文の14
遺伝子の端(mRNAの始まり)を調べるために、ニワトリの肝臓からDNAを抽出して、制限酵素で切断し、mRNAから得られたcDNAプローブと結合させた。
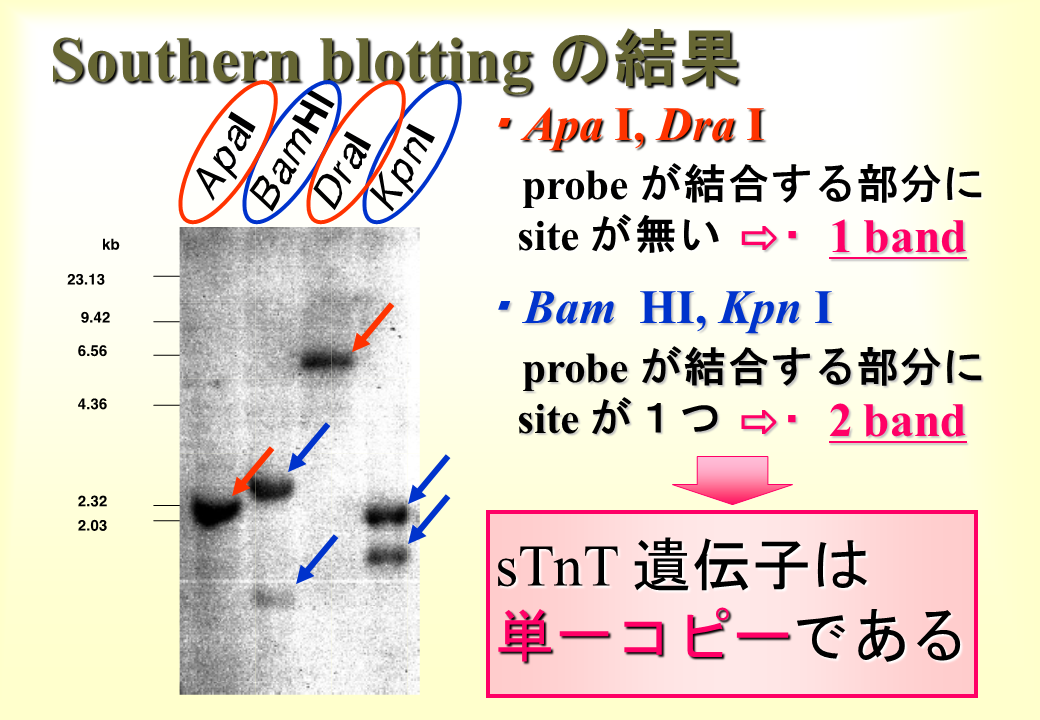
説明文の15
DNAを制限酵素ApaIとDraIで切断して、プローブと結合させた場合、1本のバンドが得られた。これは遅筋型遺伝子が1つであることを示している。制限酵素BamHIとKpnIで切断した場合には、プローブが結合する領域に制限酵素の切断部位が1つずつあるために2つのバンドがみられた。これも遅筋型遺伝子が1つであることを示している。
説明文の16
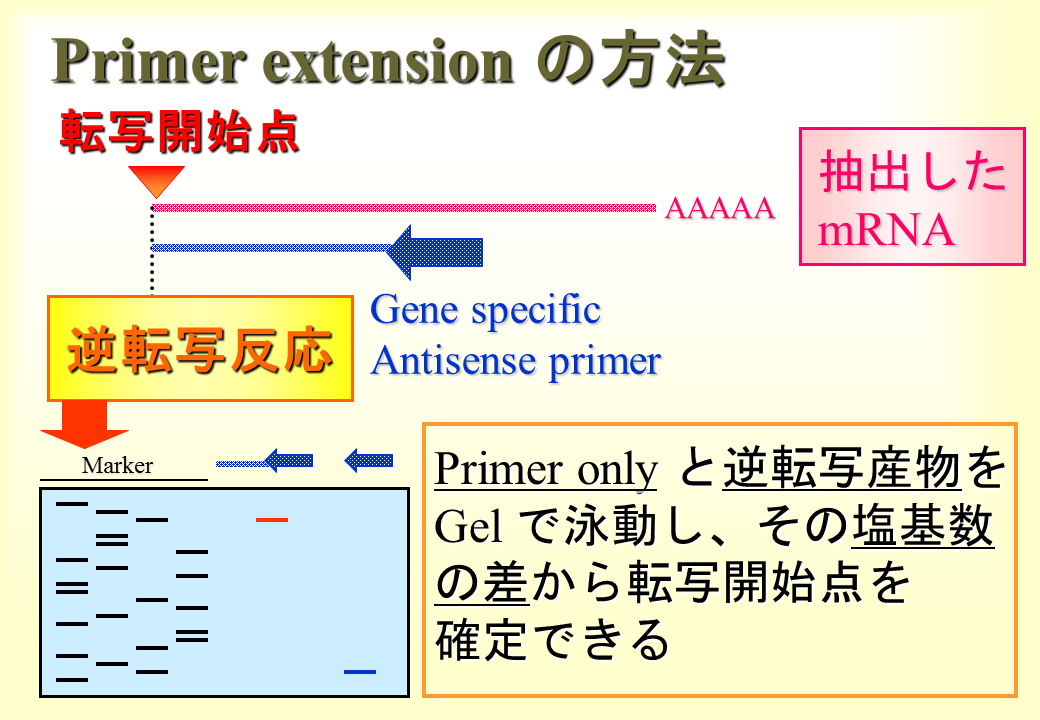
遺伝子の端(mRNAの始まり)を調べるために、ニワトリと前広背筋と肝臓からmRNAを得て、mRNAと相補的な配列をもつプライマーPE1と結合させ、逆転写酵素によりmRNAn相当する部位までcDNAを合成した。
説明文の17
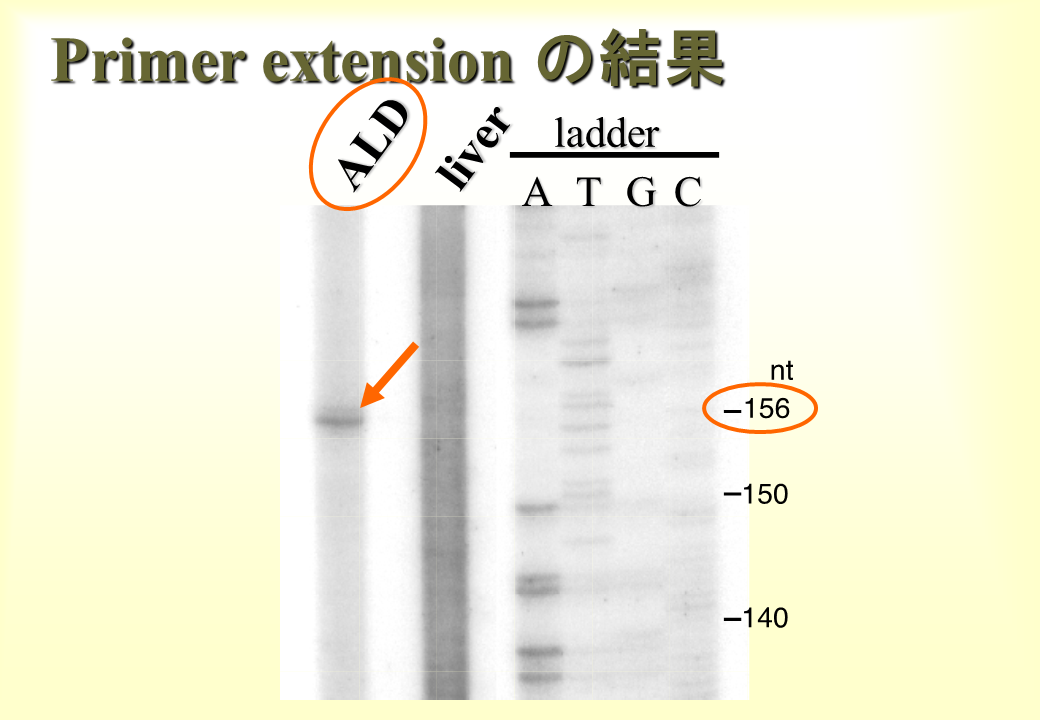
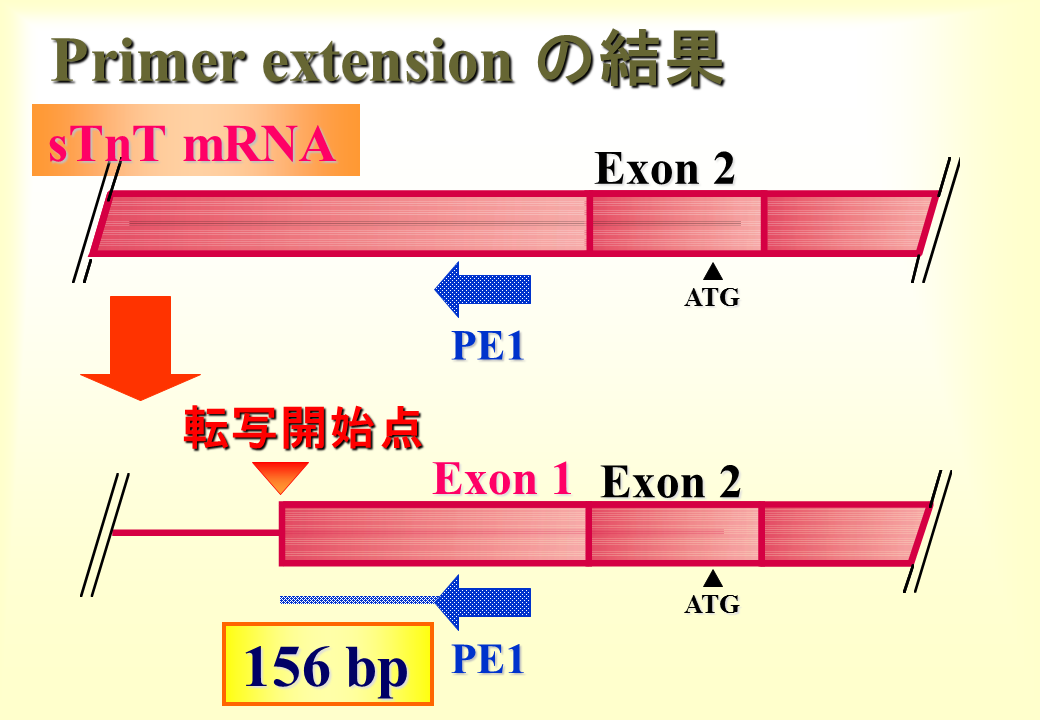
その結果、前広背筋では156 ntのバンドがみられた。それゆえプライマーから上流の遺伝子の端(mRNAの始まり)が示された。肝臓は筋肉ではないので、トロポニンTは発現しない。トロポンニンTのmRNAがないためバンドがみられなかった。
説明文の18
このようにして遺伝子の端(mRNAの始まり)、すなわちエクソン1の先端が明らかとなった。
説明文の19
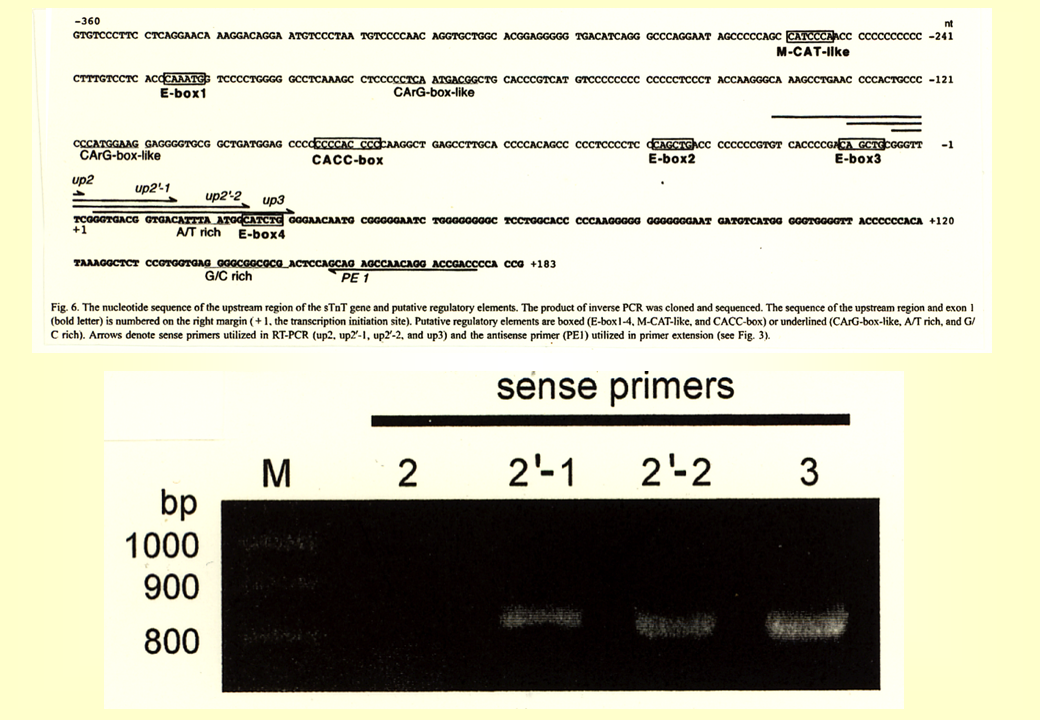
別の方法で遺伝子の端(mRNAの始まり)を調べるために、プライマー2、2‘-1、2’ -2、3各々をmRNAと結合させ、PCRを行った。先の方法で遺伝子の端(mRNAの始まり)と決定された部位が+1である。+1より上流側の部分が 多いプライマー2では、mRNAと結合できないので、増幅産物がなかった。+1より下流とかぶる領域が多くなるプライマー2‘-1、2’ -2、3を用いた場合、その順に増幅産物が多くなった。+1近辺が遺伝子の端(mRNAの始まり)であると追証された。
説明文の20
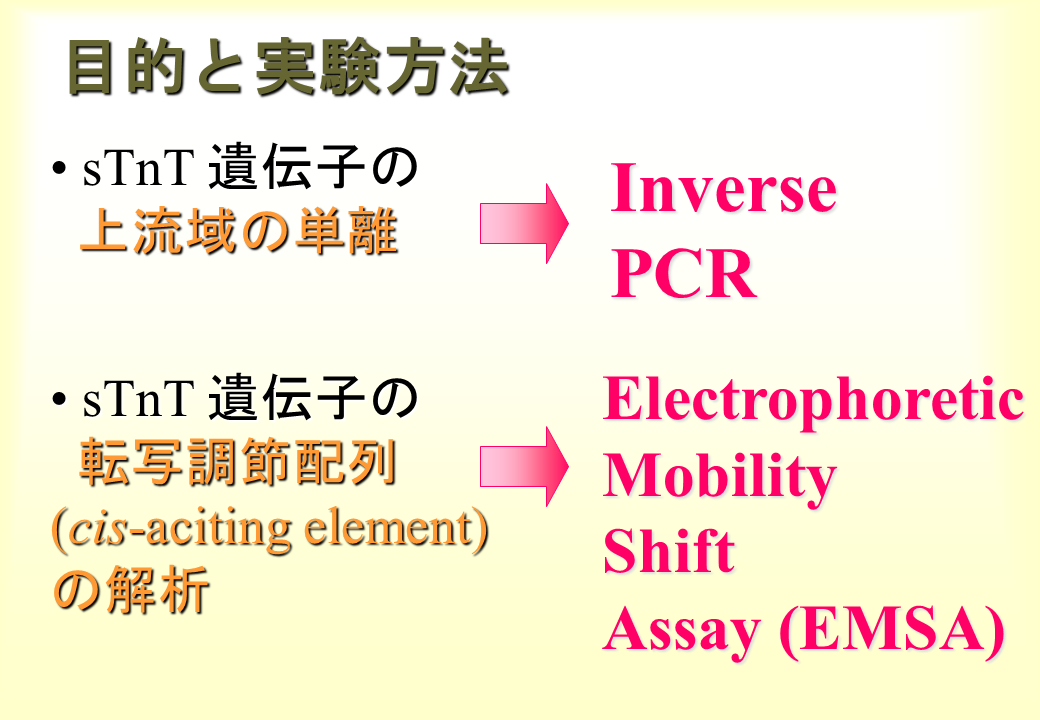
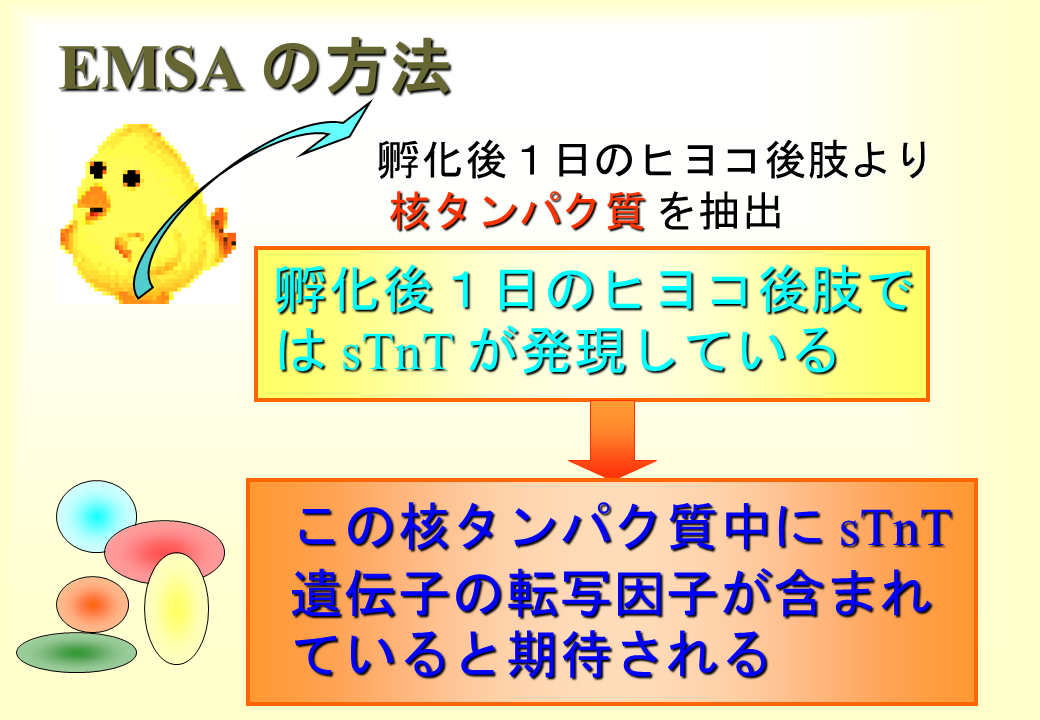
遺伝子の上流の発現調節に関わる可能性のある配列を調べるために、Inverse PCRを行った。また、実際にどの配列が発現調節に重要かを知るために、Electrophoretic Mobility Shift Assay(EMSA)を行った。

説明文の21
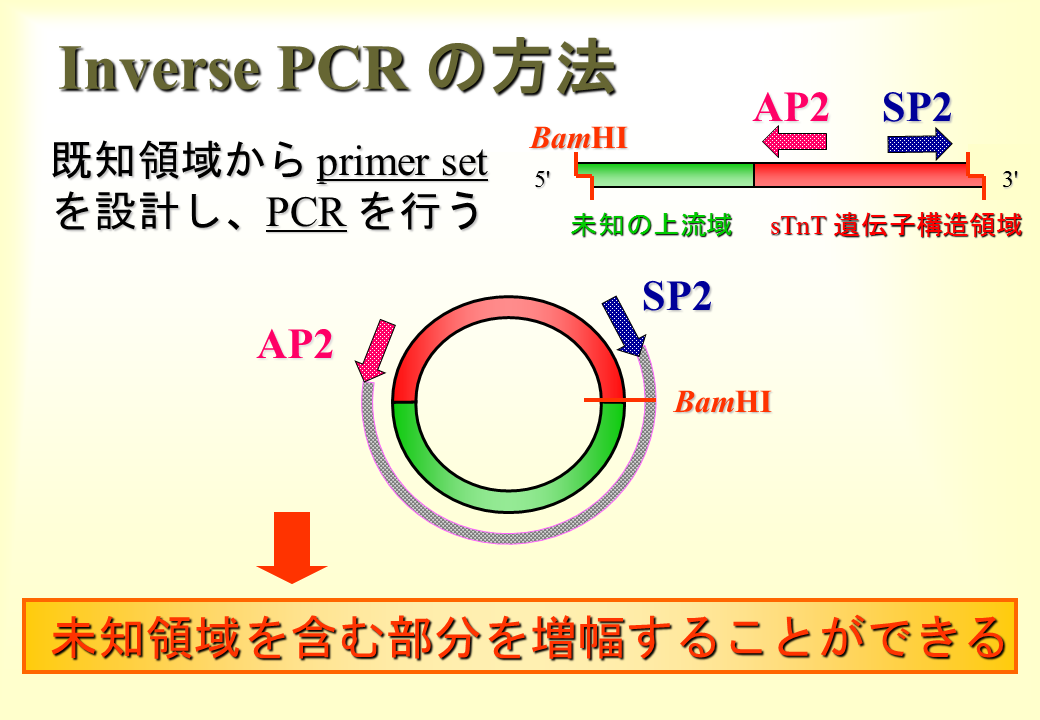
遺伝子の上流域の配列は知られていないので、プライマーを作ることができない。そのためInverse PCRを行う必要がある。肝臓よりDNAを抽出し、制限酵素BamH1でDNAを切断し(遺伝子の上流のどこにBamH1で切断される部位があるかは実際にはわかっていない)、切断した両端をつなぎ環状にした。
説明文の22
DNAが環状になっているので、プライマーAP1とSP1を用いてPCRを行うことによって、遺伝子の上流側を増幅することができる。
説明文の23
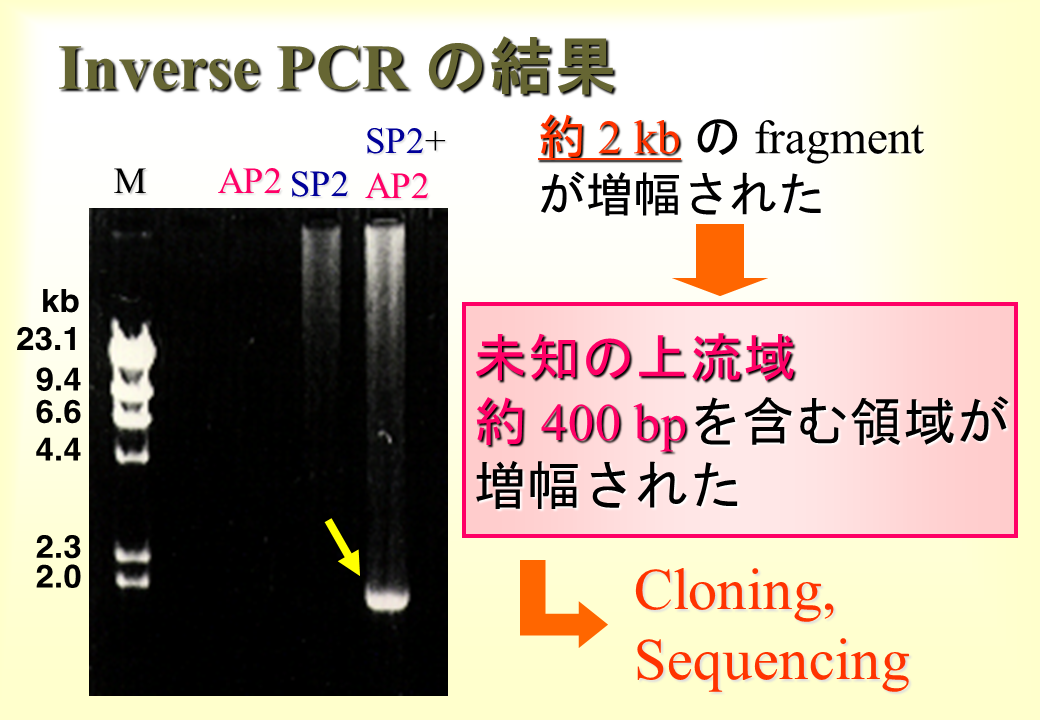
約2 kbのPCR産物が得られ、その中に約400 bpの遺伝子の上流域が含まれていた。
説明文の24
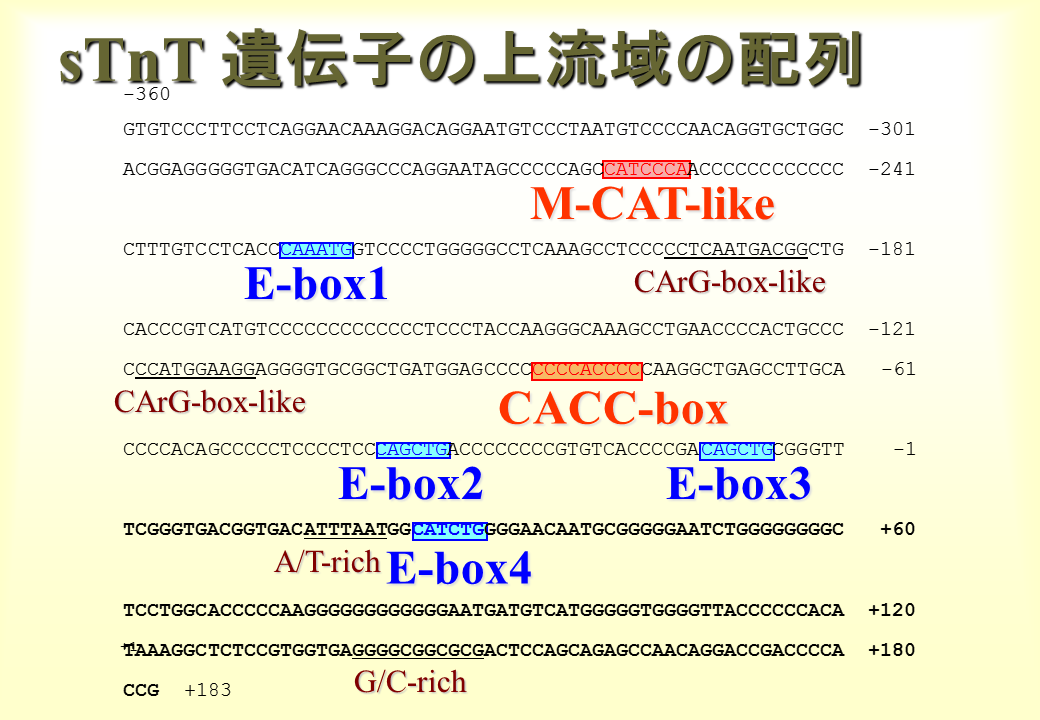
遺伝子上流の塩基配列を決めたところ、これまでの研究で遺伝子発現調節に重要であるとされた配列が複数含まれていた。
説明文の25
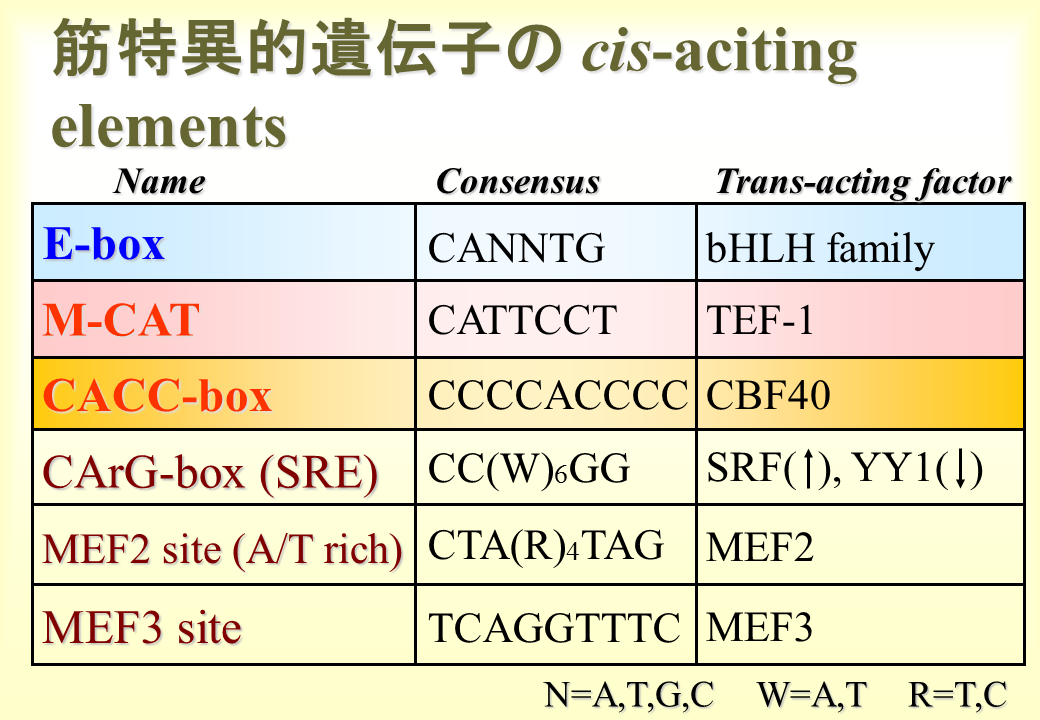
例えば、E-boxはCANNTG(NはA、G、T、Cのどの塩基でもよいことを示す)という共通の配列をもち、MyoD、Myf5、myogenin等のbHLH familyの蛋白質と結合する。このように特定の配列(cis-acting element)に結合して、遺伝子の発現調節を行う蛋白質をtrans-acting factorという。M-CATはCATTCCTという共通の配列をもち、TEF-1という蛋白質に結合する。
説明文の26
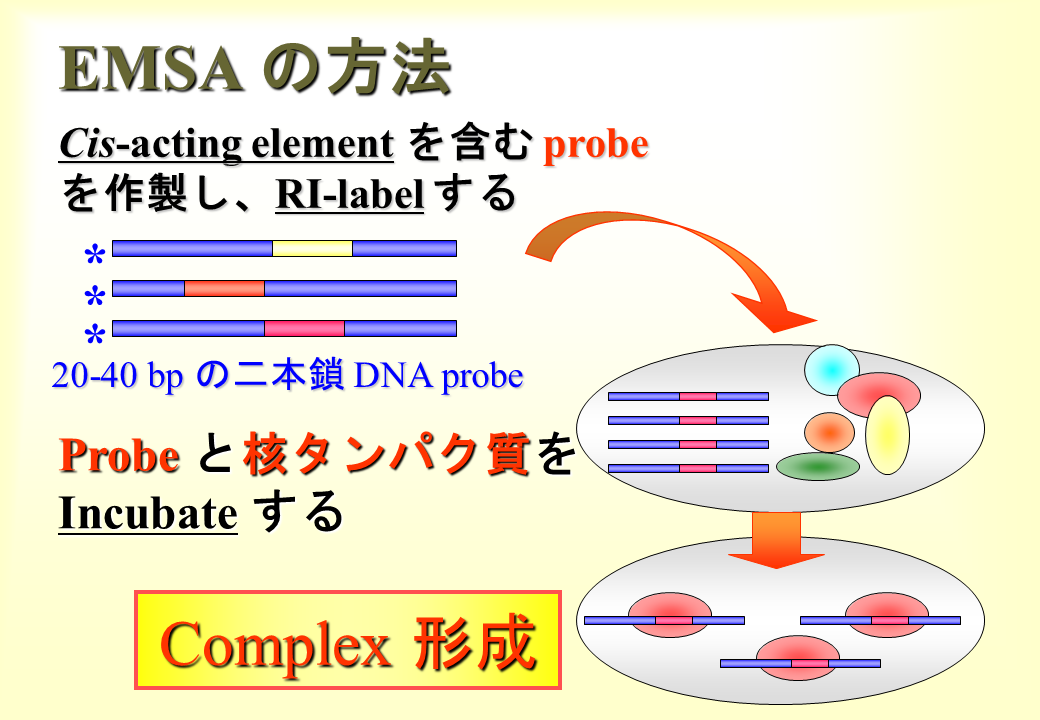
遅筋型遺伝子の上流に存在する特定の配列(E-box1〜4、M-CAT-like、CACC-box)が実際に遺伝子の発現調節に関わっているかどうかを調べるため、この配列を含むプローブを作製した。

説明文の27
6種類の特定の配列を含むプローブを作製した。赤字が遅筋型遺伝子の上流にある遺伝子の発現調節にかかわると可能性のある配列。Slow MCLはM-CAT(CATTCCT)と2塩基異なる配列(CATCCCA)をもつプローブである。
説明文の28
ニワトリの後肢の筋肉の核の中に存在する蛋白質を抽出し、プローブと後肢の筋肉の核蛋白質を結合させた。
説明文の29
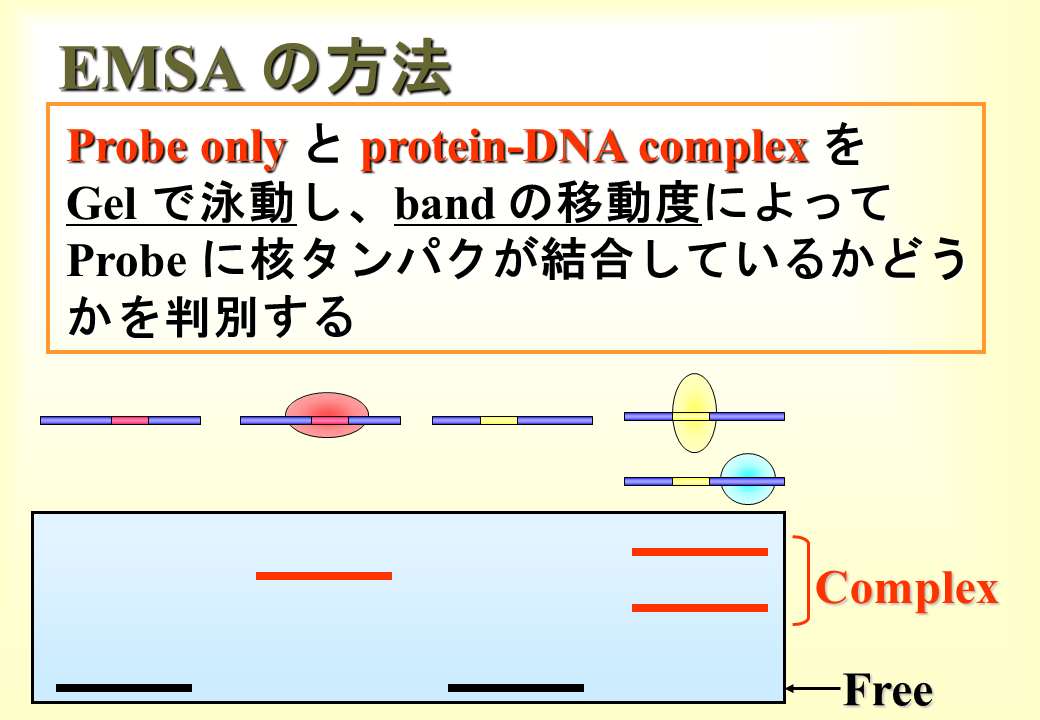
これを電気泳動すると、プローブに核蛋白質が結合していない場合は、プローブのサイズ(分子量)に従って移動する。一方、結合している場合は、分子量が大きくなり、移動度が遅れる。そのようにして特定の配列に核タンパク質が結合しているか判定する。この方法をEMSAという。
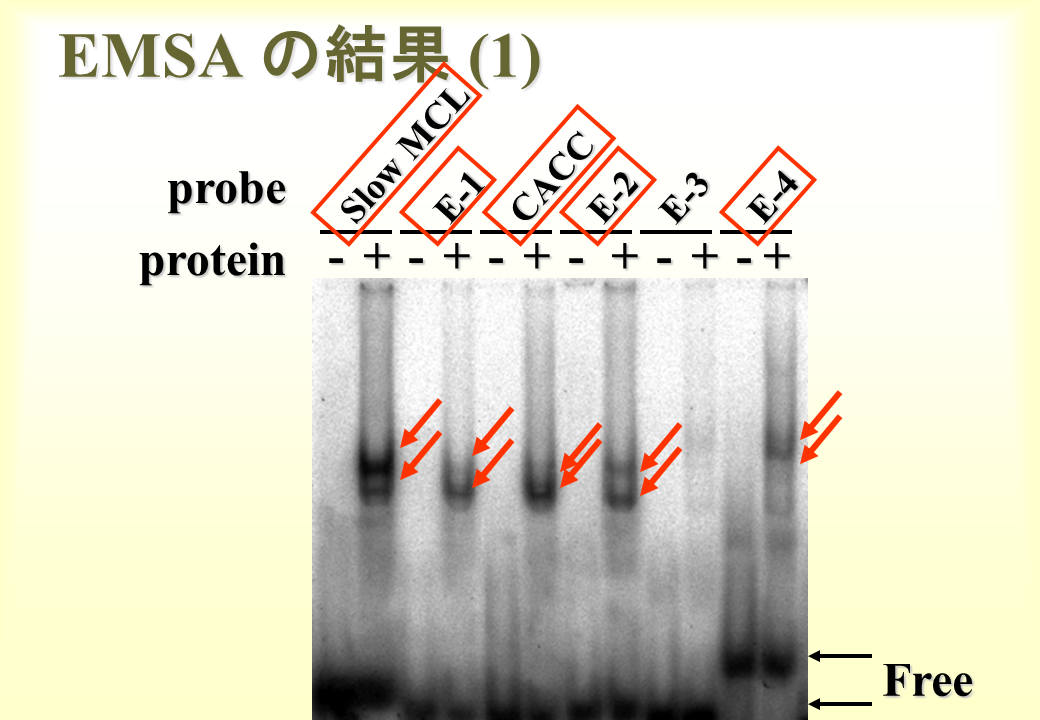
説明文の30
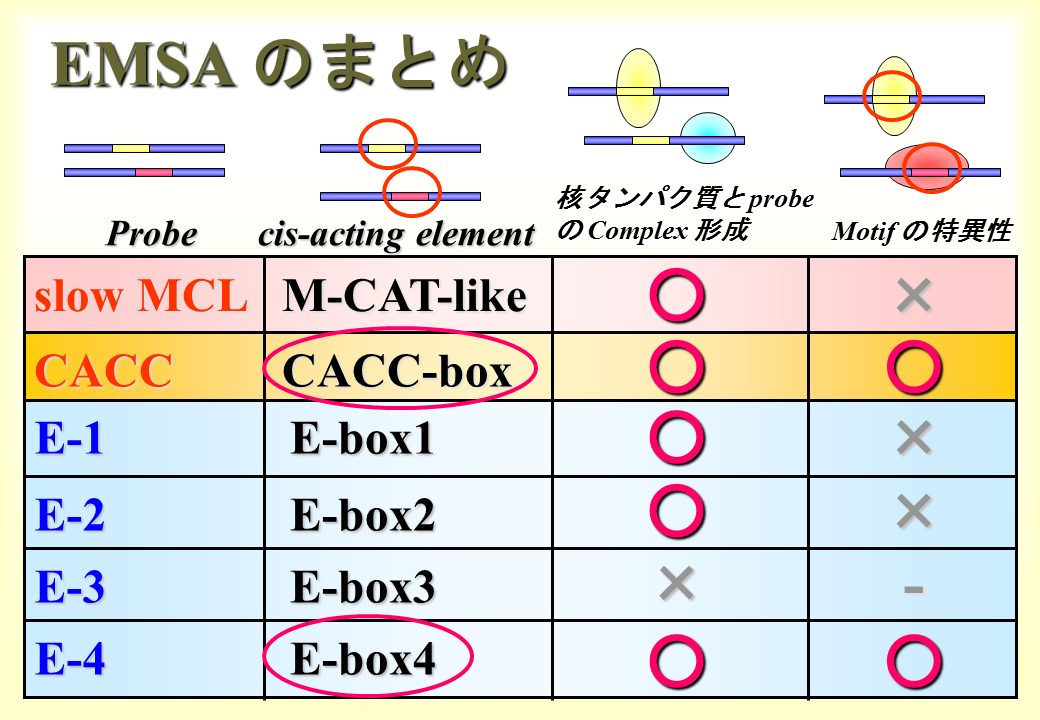
E-box1〜4、Slow MCL(M-CAT-like)、CACC-boxの配列を含むプローブを核蛋白質と結合させると、E-3以外すべてが核タンパク質と結合して移動度が遅れた。E-3は遅筋型トロポニンT遺伝子の発現調節に関わっていないことが明らかとなった。
説明文の31
本当にE-box1、2、4、Slow MCL(M-CAT-like)、CACC-boxの配列と核タンパク質が結合しているか確認するために、ラベルしたプローブに対して、ラベルしていないプローブを10倍量ならびに100倍量用いて、EMSAを行った。
説明文の32
過剰量のラベルしていないプローブに核タンパク質が結合すると、ラベルしたプローブに結合しうる核タンパク質の量は少なくなる(バンドが薄くなる)はずである。
説明文の33
また、特定の配列の一部を変えたmutantプローブを作製して、ラベルしたプローブに対して、ラベルしていないプローブを10倍量ならびに100倍量用いて、EMSAを行った。ラベルしたプローブは本来の(変化させていない)配列をもっている。

説明文の34
E-box1、2、4、Slow MCL(M-CAT-like)、CACC-boxの配列の一部を変えて、mutantプローブを作製した。
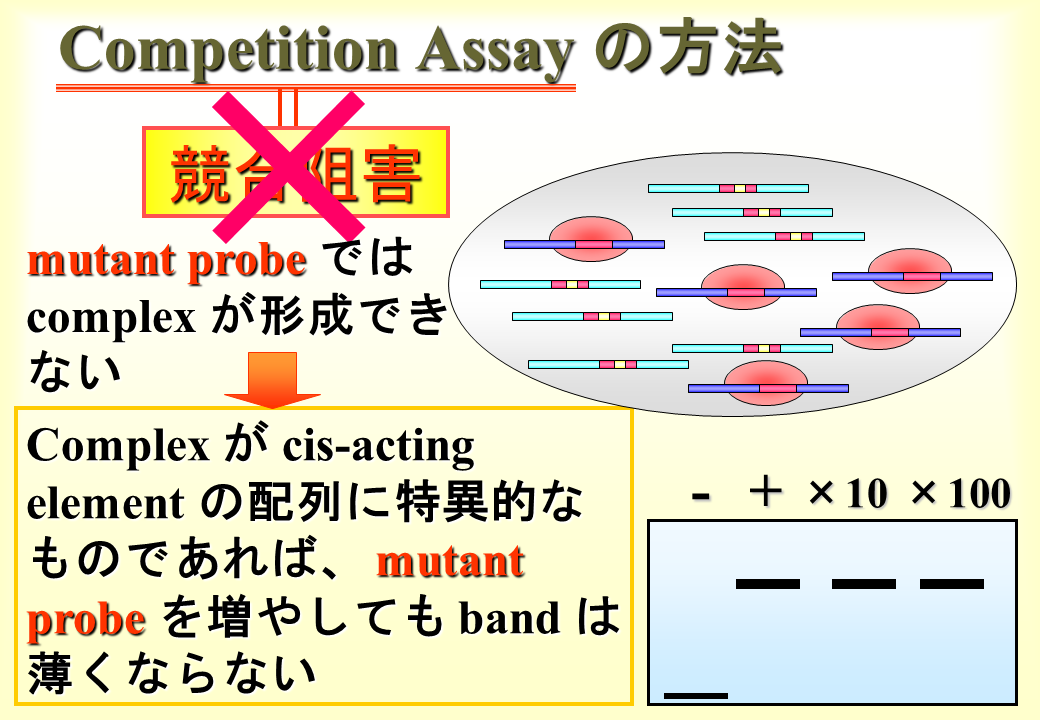
説明文の35
特定の配列をもつプローブに厳密に核タンパク質が結合するならば、配列の一部を変えたmutantプローブには結合できないはずである。したがって、ラベルしていないmutantプローブをいくら大量に加えても、そのプローブには結合できないのだから、本来の配列をもつラベルしたプローブと核蛋白質との結合に影響を与えない。すなわち、バンドは薄くならない。
説明文の36
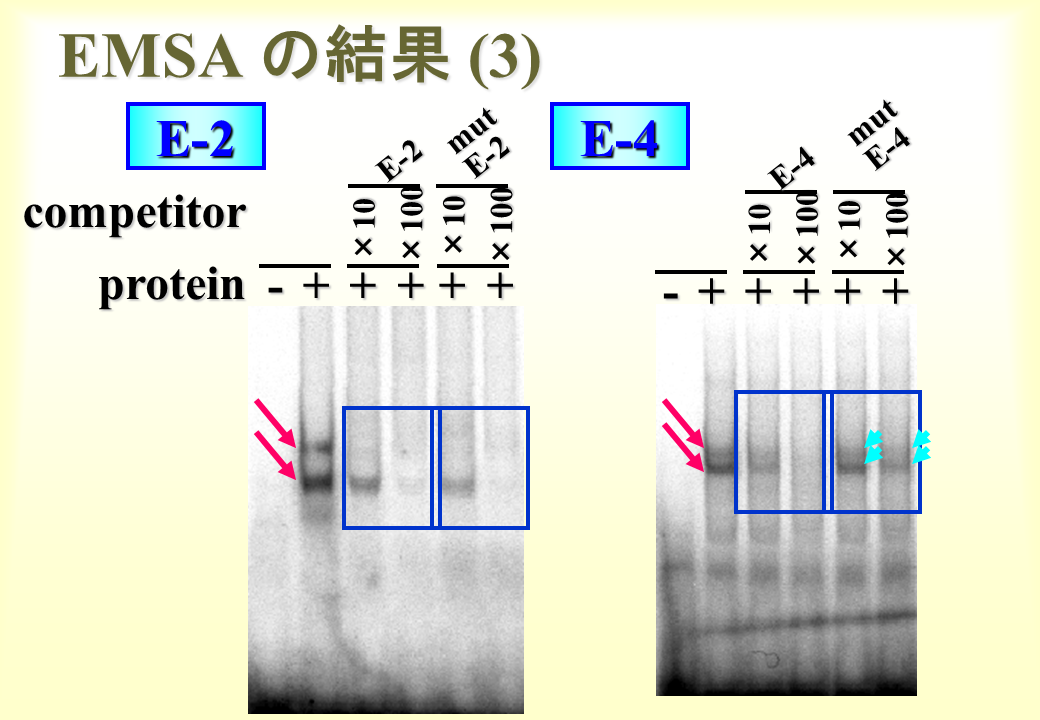
E-2でもE-4でも、過剰量の本来の配列をもつプローブを過剰量加えた場合、バンドの濃さが薄くなった。しかし、E-2では、mutantプローブを過剰量加えてもバンドが薄くなったので、本来の配列以外のプローブの他の部分に核タンパク質が結合した可能性がある。一方、E-4では、mutantプローブを過剰量加えてもほとんどバンドが薄くならなかったので、本来の配列に核タンパク質が結合していることが明らかとなった。
説明文の37
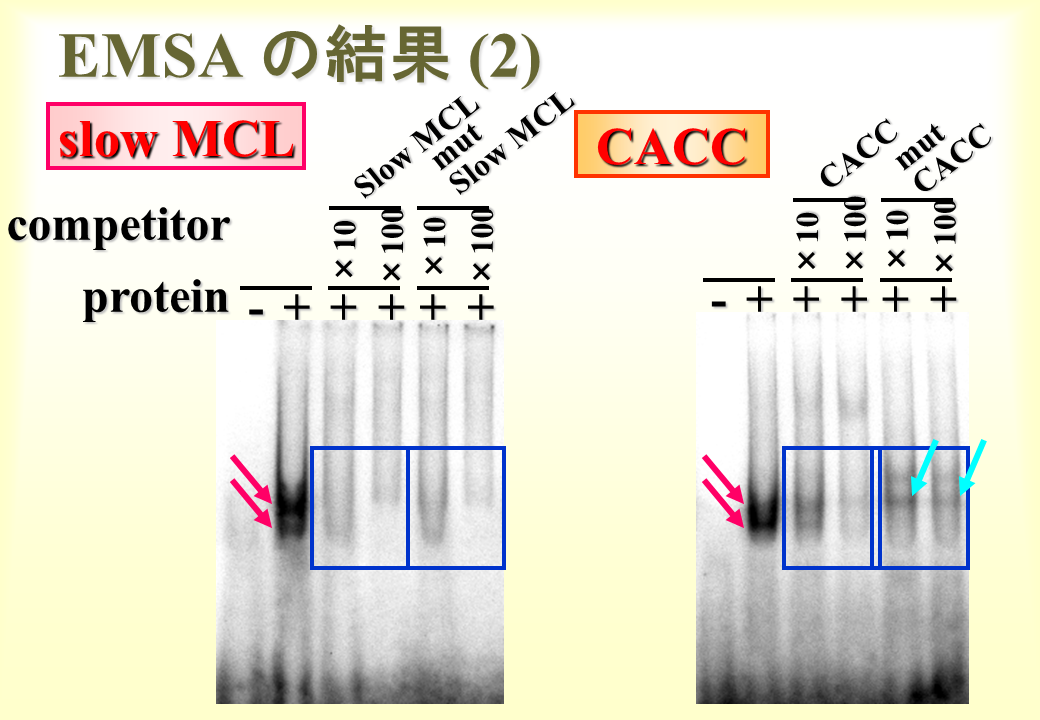
Slow MCLに関しては、E-2と同じでmutantプローブを過剰量加えてもバンドが薄くなったので、本来の配列に核蛋白質が結合したのではないことがわかる。CACC-boxに関しては、E-4tと同じでmutantプローブを過剰量加えてもほとんどバンドが薄くならなかったので、本来の配列に核タンパク質が結合していることがわかる。
説明文の38
まとめると、EMSAによる実験から、CACC-boxとE-box4が遅筋型トロポニンT遺伝子の発現調節にかかわる可能性が高くなった。

説明文の39
遅筋型トロポニンTでは、CACC-boxとE-box4が遺伝子の発現調節にかかわる可能性があるが、これまでの研究から、遅筋型の遺伝子で働く配列としては、SURE(CACC-box、MEF2、 E-box)、MEF-2、calucinerin-depedant NFATが知られている。
説明文の40
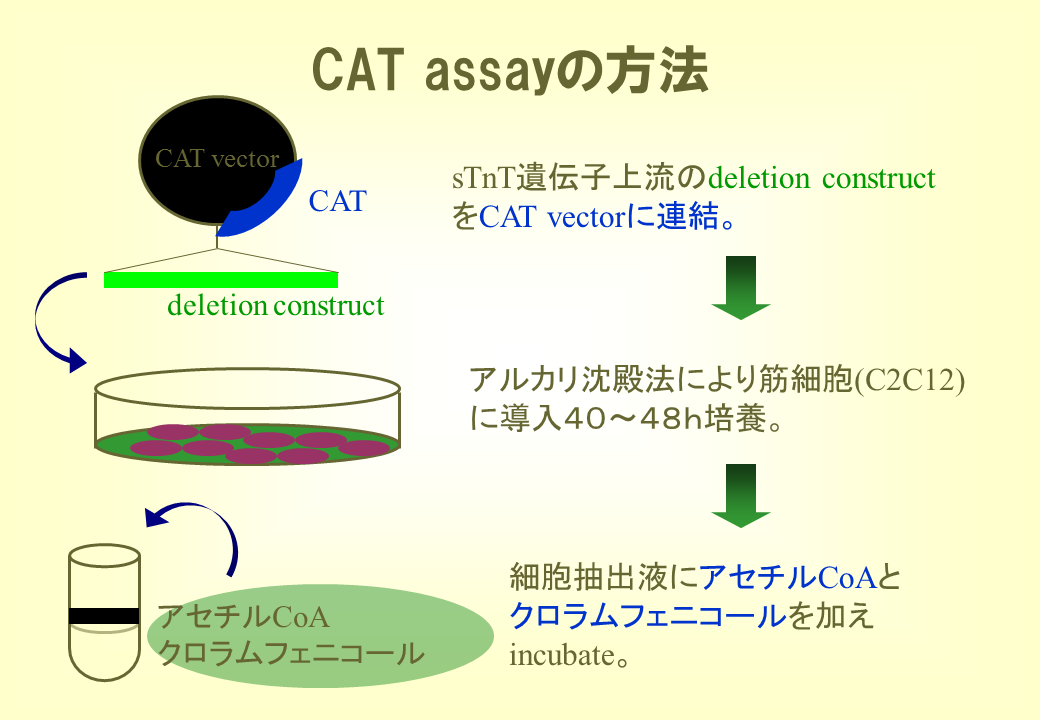
次に、本当にCACC-boxとE-box4が遺伝子の発現調節にかかわるか調べるために、CAT assayを行った。遅筋型トロポニンT遺伝子の発現調節にかかわると考えられる上流域を徐々に削っていき、これを大腸菌のCAT遺伝子の上流に連結する。この構築物(遅筋型トロポニンT遺伝子の上流域を挿入したCAT vector)を遅筋から得た培養細胞に取り込ませ、細胞を培養する。その後、CAT遺伝子の発現を調べる。
説明文の41
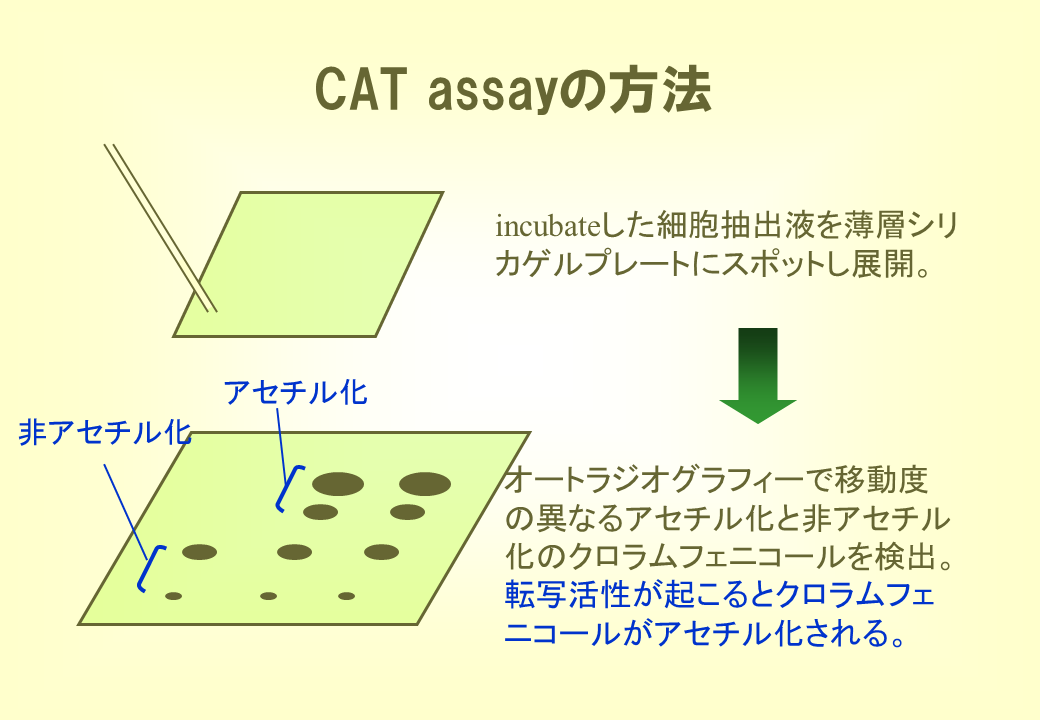
CAT(クロラムフェニコールアセチル化酵素)遺伝子が働くと、基質のクロラムフェニコールがアセチル化され、薄層クロマトグフィーによって、アセチル化されたものが検出されるようになる。
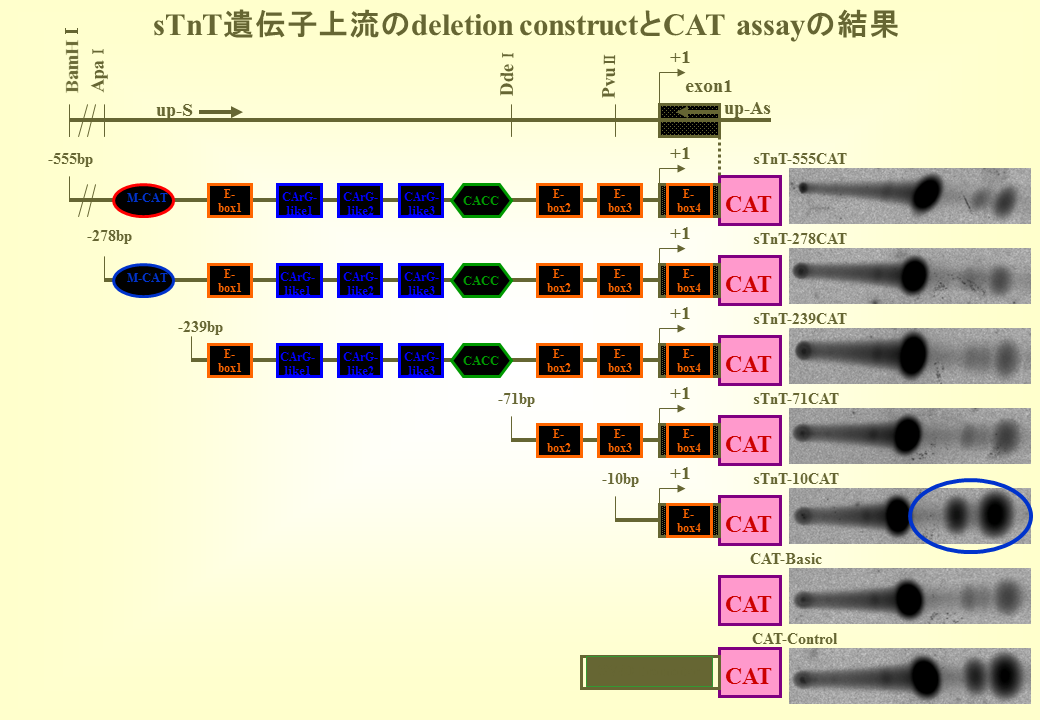
説明文の42
遅筋型トロポニンT遺伝子の上流域を徐々に削っていき、E-box4だけを含む短い領域をCAT遺伝子に連結した時、明確にクロラムフェニコールがアセチル化された。E-box4も削ってしまうと、クロラムフェニコールがアセチル化されなくなった。したがって、確かにE-box4は遅筋から得た培養細胞の中で、その下流の遺伝子を活性化することがわかった。一方、CACC-boxはあってもなくても、下流の遺伝子の発現は変化しなかった。一番下のSV40 Promoterはどんな細胞でも下流の遺伝子を活性化させるので、いわゆるポジティブコントロールである。

説明文の43
基部はもともと速筋であったが、徐神経してもそのまま速筋であることがわかり、神経支配はE-box4を含んでいる遅筋型トロポニンT遺伝子の上流域が、CAT遺伝子を活性化するこができなかったのはなぜか?このことはE-box4だけで全てが説明できるのではなくて、E-box4の上流にある何らかの調節領域も関係している可能性を示している。とにかくこの実験はまだ1度しか成功していない。再現性があるか検討しなければならない。
説明文の44
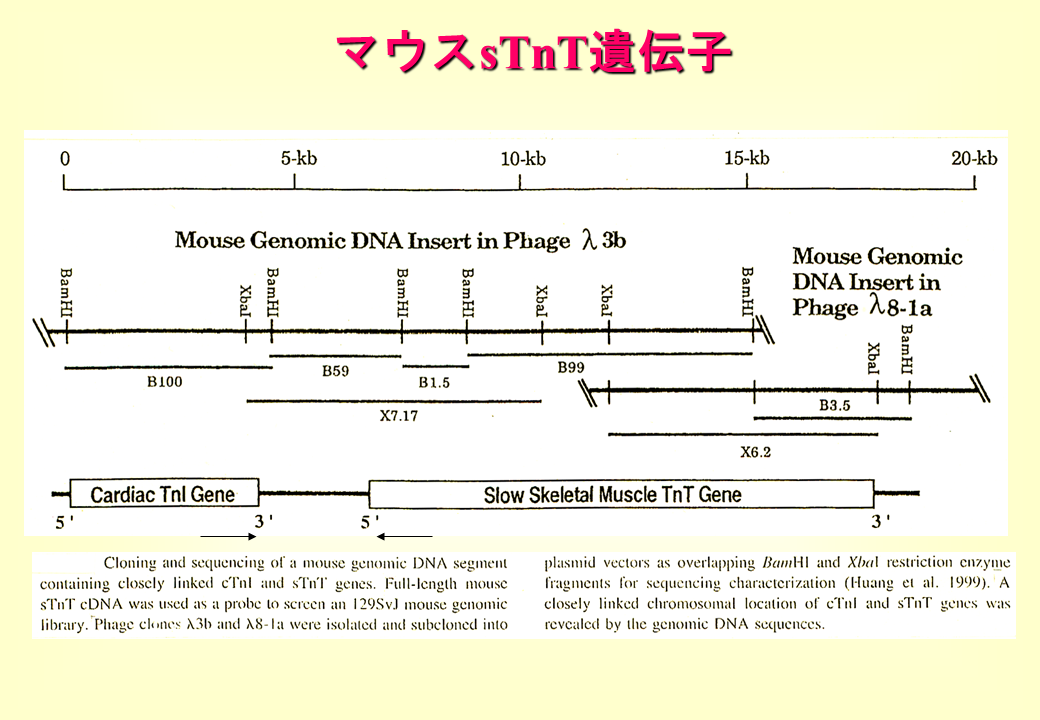
マウスでは心筋型トロポニンI遺伝子と遅筋型トロポニンT遺伝子が並んでいるらしい。
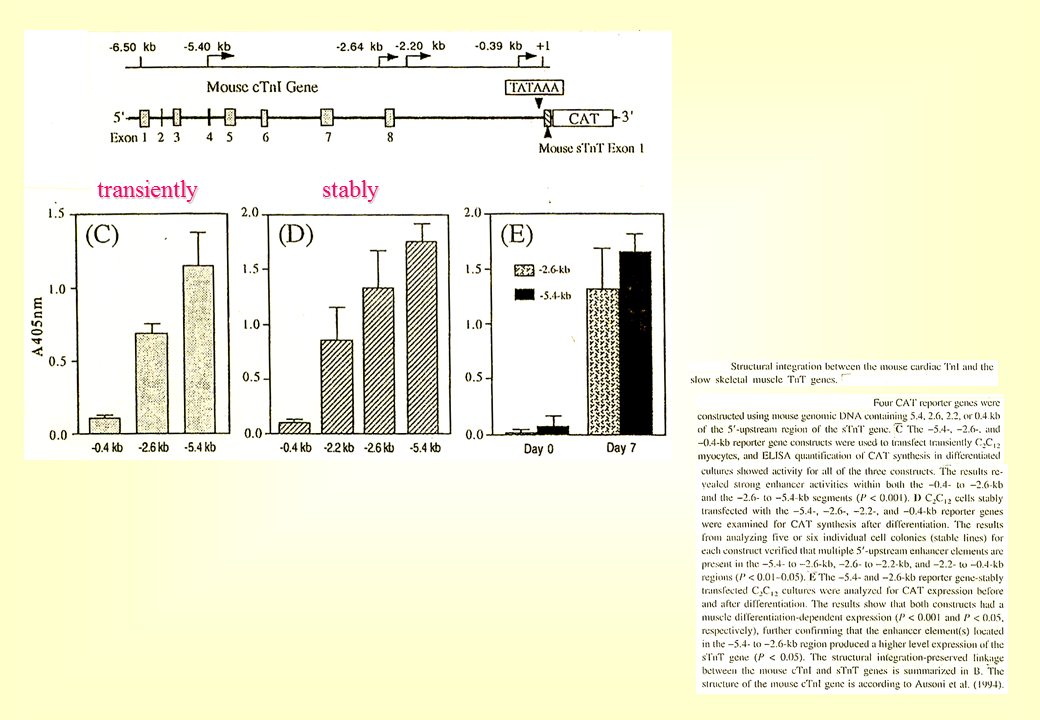
説明文の45
また、遅筋型トロポニンT遺伝子の発現の調節が、心筋型トロポニンI遺伝子と遅筋型トロポニンT遺伝子との間の領域だけでなく、心筋型トロポニンI遺伝子内の領域もかかわっている可能性を示している。ニワトリでも心筋型トロポニンI遺伝子と遅筋型トロポニンT遺伝子は並んでいるのだろうか、また、マウスと同じように遺伝子発現が調節されいるのであろうか、調べる必要がある。
